Un equipo interdisciplinario liderado por el Dr. Rodolfo Porasso, docente investigador del Departamento de Física y del IMASL (UNSL-Conicet) estudia el comportamiento de membranas celulares frente a diversas situaciones problemáticas. Analizan mediante la simulación computacional, cómo reacciona esa capa superficial de las células frente al ingreso de fármacos, contaminantes, sustancias nocivas, o incluso, diferentes tipos de virus.
Del universo biológico, a este grupo de investigación le interesan las células y particularmente la membrana que separa el interior del exterior de la misma. Esta capa presenta particularidades de enorme complejidad, tanto en su composición como en su comportamiento frente a distintos compuestos o condiciones.
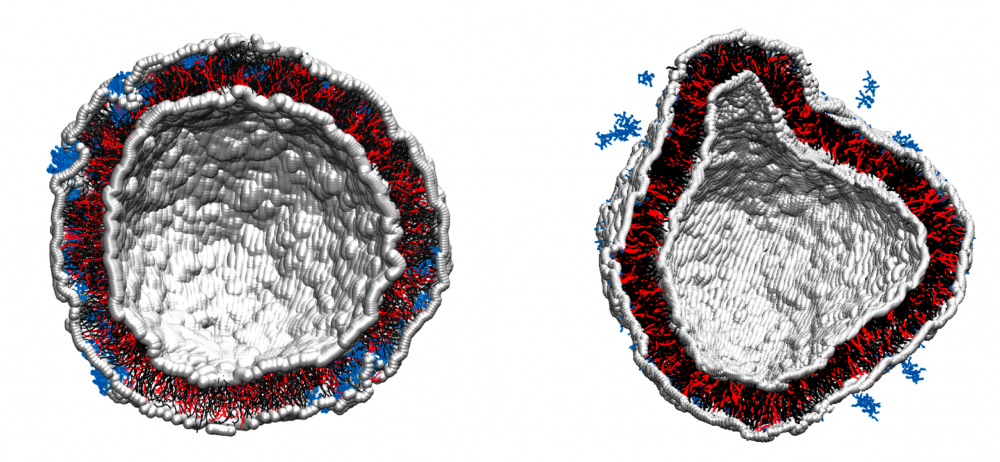
«La membrana celular es algo extremadamente complejo, tiene muchos compuestos químicos, lípidos, colesterol, proteínas etc. Lo que hacemos es identificar, por ejemplo, los lípidos, que a su vez tienen una característica peculiar ya que a una parte le gusta estar en contacto con el agua y la otra parte no. Desde la simulación creamos una especie de baldosa que representa esa membrana, le asignamos las características específicas como la carga, el tamaño, constantes eléctricas y así simulamos los distintos sistemas», explica Porasso.
En la simulación computacional trabajan con distintos tipos de moléculas que penetran la membrana celular y allí pueden observar y analizar cómo se lleva a cabo ese procedimiento, e identificar cuáles son sus particularidades. «Tratamos de ver desde el punto de vista físico, cuál es el mecanismo en que los distintos fármacos pueden atravesarla. Empezamos con moléculas simples como la benzocaína que tiene 12 átomos, luego lo hicimos con el ibuprofeno, otra molécula también de unos pocos átomos», señala.
Actualmente estudian cómo funcionan los antibióticos que son moléculas mucho más grandes que con las que iniciaron la investigación. Porasso subraya que no tratan de explicar el mecanismo por el cual estos antibióticos curan, sino el mecanismo por el cual ingresan a la célula.
El equipo está integrado por físicos, biólogos moleculares, químicos, bioquímicos y farmacéuticos quienes aportan una visión diferenciada de los problemas que trabajan en el Proyecto. «Es un grupo bastante interdisciplinar y es totalmente transversal, todos colaboramos y vemos el mismo problema, pero cada cual, con su enfoque, y ahí es donde más enriquece la resolución del problema, porque a los físicos nos interesan los cambios de energía libre, fuerzas impulsoras, cambio de entropía. Para los bioquímicos es mejor estudiar la concentración y los biólogos moleculares se enfocan en las células. Cada cual tiene su mirada particular, y ahí es donde nos enriquecemos todos», agrega.
Los conocimientos a los cuales se arriban en este Proyecto permiten dotar de información relevante a la industria farmacológica, a laboratorios y otros equipos de investigación con los que trabajan colaborativamente. «Nosotros hacemos ciencia básica, pero estas simulaciones aportan datos que posibilitarían hacer fármacos que sean más eficientes, optimizando la dosis al mejorar el mecanismo de inserción en la célula».
Porasso explica que vienen trabajando con equipos del Instituto Multidisciplinario de Investigaciones Biológicas (IMIBIO UNSL-Conicet), con el Instituto Pasteur de Uruguay, las Universidades de Murcia y Valencia de España y grupos de investigación de Hungría. Estos nexos resultan muy significativos para el proyecto toda vez que les aporta la parte de la experimentación, la posibilidad de verificar en un experimento, los procesos simulados digitalmente. «La fuente de nuestros datos experimentales está en general, en Europa. Ellos hacen los experimentos y por suerte, hay una correspondencia muy grande entre los datos experimentales y nuestras simulaciones. Lo hacemos a doble ciego, para que no haya sesgo», sostiene.
Como todo proyecto de investigación, éste tiene diversas líneas de trabajo. En una de ellas, un estudiante doctoral pudo desarrollar después de dos (2) años de arduo trabajo, una simulación de un virus porcino. Uno de los mayores desafíos de este equipo es empezar a trabajar con distintos tipos de virus. «Intentamos desarrollar desde la simulación, algo que logre romper ese virus. Por lo pronto logramos construirlo y ya eso es un montón, construir un virus desde el punto de vista de las simulaciones requiere muchísimo esfuerzo, ahora veremos cómo lo podemos atacar», anticipa.
Ciencia y vocación
La Facultad de Ciencias Físico Matemáticas y Naturales tiene una fuerte trayectoria científica. Hoy alberga muchos proyectos de investigación que dirigen científicos y científicas que también transitaron sus pasillos como estudiantes, y en esa etapa formativa, tuvieron referentes que contagiaron su pasión por la investigación.
«Nuestra carrera es una carrera chica. Cuando yo era estudiante los profesores que tenía nos conocían a todos, no hacía falta ni poner el nombre a la hoja, que ya sabía de quién era la letra. Eso formó un vínculo muy estrecho entre los docentes y los estudiantes (…) Dentro del Departamento de Física que científicamente es muy fuerte, hubo docentes como Giorgio Zgrablich, que es casi un prócer en la investigación en Argentina. Uno lo veía a él, cómo trabajaba, la pasión que tenía por la investigación y te contagiaba ese entusiasmo. Quien me formó a mí fue Julio Benegas, que dirigía el otro grupo grande de investigación y trabajar con ellos, haber sido estudiante de ellos, contagia. Ahora que estamos de este lado, uno trata de hacer lo mismo con los estudiantes actuales».
Nota: FCFMyN | Unidad de Cultura Científica más Innovación (UCC+i)

