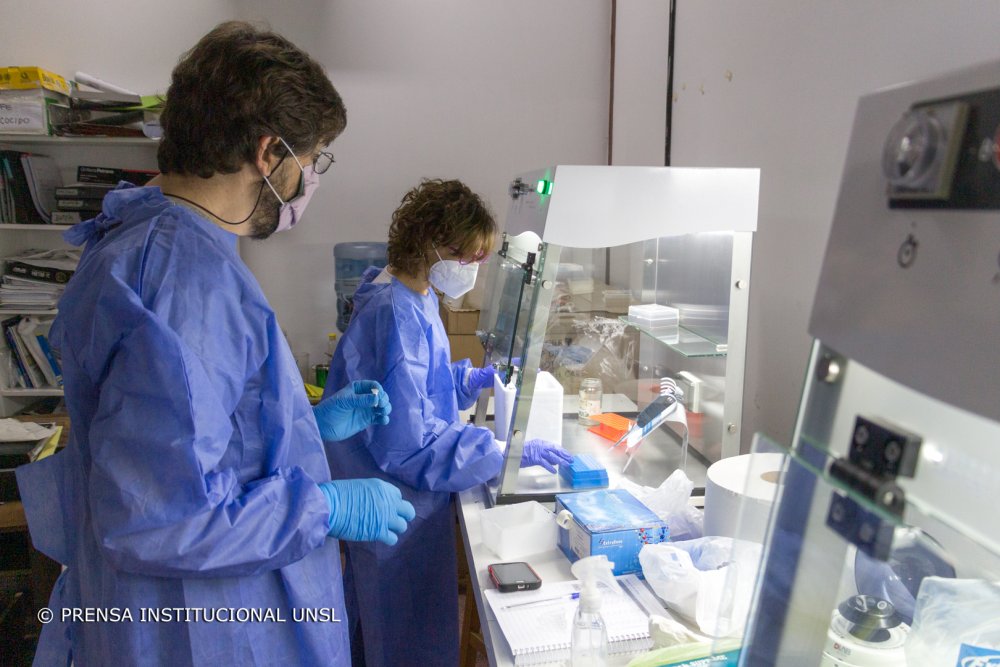
El Dr. Maximiliano Juri Ayub y la Dra. Jimena Manzur buscan optimizar un test de saliva para la detección de asintomáticos e identificar las variantes de interés epidemiológico del SARS-Cov 2. En este sentido, los investigadores del Área de Biología Molecular del Departamento de Biología de la Facultad de Química, Bioquímica y Farmacia (FQByF), dialogaron con Noticias UNSL sobre sus avances.
En el marco de un Proyecto Institucional financiado por el Ministerio de Ciencia, Tecnología e Investigación y la Universidad Nacional de San Luis (UNSL), los científicos proponen implementar, adaptar y diseñar estrategias de Biología Molecular que puedan contribuir con herramientas diagnósticas y/o epidemiológicas durante la pandemia por Covid-19.
¿Cuál es el objetivo de su trabajo?
Creemos que una de las funciones del sistema de Ciencia y Tecnología durante la pandemia debe ser ampliar el menú de opciones disponibles para que las autoridades sanitarias tomen las decisiones pertinentes. En ese marco, a partir del estudio de una enorme cantidad de información científica generada en corto tiempo en todo el mundo, surgen diferentes propuestas, las cuales hay que diseñar, ensayar, validar y poner a disposición en caso que funcionen como se espera.
Test de saliva para la detección de Covid-19
¿Qué nos pueden decir sobre la posibilidad de realizar tests de detección en saliva?
El test diagnóstico posee tres (3) etapas: la toma de muestra, la extracción del material genético viral y, finalmente la detección específica de genes virales. Cada etapa tiene sus particularidades y complicaciones, sobre las cuales se ha trabajado mucho para simplificarlas y hacerlas disponibles masivamente. En particular, las dos (2) últimas etapas son hoy mucho más sencillas y rápidas que al comienzo de la pandemia. La toma de muestra, sin embargo, se ha modificado poco en esencia. Demanda personal entrenado, es laboriosa, requiere el uso constante de material de protección, etc. Desde un inicio se planteó el test a partir de saliva como una opción, aunque los resultados iniciales de diversos trabajos eran poco consistentes. Hoy en día existe cierto consenso de que la detección del virus a partir de saliva puede ser muy útil, sobre todo como un método de vigilancia epidemiológica en personas asintomáticas.
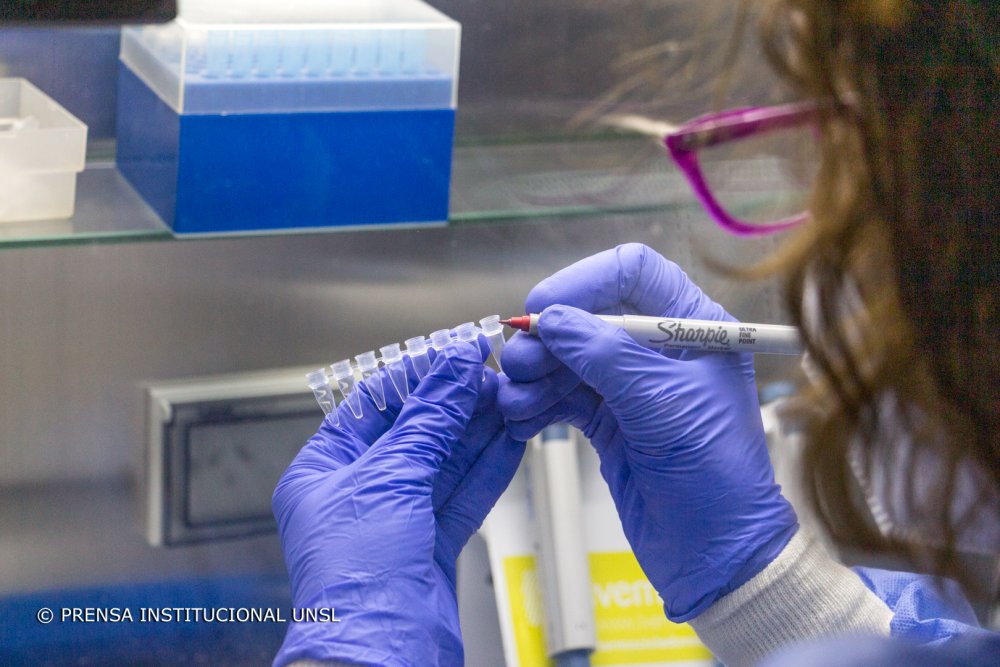
¿Este test de saliva está orientado hacia un público específico?
Como decíamos antes, nuestra función es mejorar, adaptar o proponer herramientas. La decisión de cómo y cuándo aplicarlas corresponde a las autoridades sanitarias, que analizan la situación desde una perspectiva más amplia. Se postula en principio como un método para análisis de asintomáticos, mediante una toma de muestra más sencilla y amigable, tanto para el personal de salud como para el paciente. En principio se piensa con fines de vigilancia epidemiológica más que de diagnóstico clínico.
¿Cómo sería su forma de detección?
El protocolo de extracción de material genético y detección es idéntico al empleado actualmente para los hisopados. Esto es otra ventaja porque no requiere modificar protocolos en funcionamiento.
¿Cuándo se podría comenzar a implementar?
El primer paso, como en cualquier posible modificación de una metodología diagnóstica, es realizar una comparación entre el desempeño de la técnica de referencia (en este caso el hisopado) y la modificada. En particular, el parámetro clave a conocer es la sensibilidad. Varios trabajos científicos recientes muestran una sensibilidad comparable a la metodología estándar, pero eso debe ser validado localmente.
Variantes de interés epidemiológico
¿Cómo están trabajando para reconocer las cepas de SARS-Cov 2?
Uno de los últimos desafíos que han surgido es la aparición de las llamadas variantes de interés epidemiológico. Estas consisten de linajes del SARS-Cov 2 que poseen, aparentemente, características preocupantes (en particular mayor transmisibilidad y/o letalidad, o eventualmente escape al efecto protector de las vacunas). Lamentablemente, en el país está confirmada la circulación comunitaria de estas variantes.
¿En qué se distingue una cepa de la otra?
Las cepas o variantes se caracterizan por cambios (llamadas mutaciones), en general muy pequeñas, en la información genética del virus, respecto de la cepa original. Ocasionalmente, algunos de esos cambios, pueden generar características preocupantes (mayor infectividad, mayor transmisibilidad, letalidad, escape del sistema inmunológico).
¿Cómo es el procedimiento para detectar estas variantes?
El procedimiento consiste en secuenciar, mediante métodos caros y laboriosos, el genoma completo o partes del genoma del virus, a partir de muestras de cada paciente que se sospeche puede contener la variante (por ejemplo, por proceder de una región con alta circulación de dicha variante). Si bien se ha ampliado la capacidad de secuenciación en el país, este procedimiento no puede realizarse a cada muestra, sino que se realiza sobre muestras seleccionadas. Por lo tanto, junto a colegas de otras provincias, estamos diseñando estrategias por metodologías más sencillas, como PCR, que podrían implementarse para todas las muestras, y detectar «candidatas» para la secuenciación, descartando la mayoría de las sospechosas en un paso de tamizaje previo. Esto es posible gracias al hallazgo (a través del análisis bioinformático de miles de genomas virales) de un laboratorio de la Universidad de Yale, en Estados Unidos. Las variantes de interés epidemiológico comparten entre sí algunas mutaciones, que además son más grandes que las esperadas (ausencia de pequeñas regiones del genoma en lugar de cambios puntuales). Estos cambios de magnitud mayor podrían, en principio, ser detectados por PCR de manera más sencilla.
¿Cuál es la importancia de conocer el origen de cada cepa?
Lo sustancial no es el origen, sino las características de cada variante. Es importante, sin embargo, notar que las variantes de interés epidemiológico surgieron, no casualmente, en regiones de alta circulación viral (Inglaterra, Brasil, Sudáfrica). Por lo tanto, la mejor manera de evitar el surgimiento de nuevas variantes preocupantes es disminuir la circulación viral, principalmente a través de medidas de distanciamiento y vacunación masiva.

